在细胞生物学研究中,理解蛋白质之间的相互作用如同解开生命奥秘的关键钥匙。然而,传统的蛋白质互作研究方法如免疫共沉淀(Co-IP)往往存在局限性,无法捕捉瞬态或弱相互作用。近年来,邻近标记技术崭露头角,以其高时空分辨率成为研究蛋白质互作和细胞器蛋白质组的明星工具。本文将带您深入了解APEX2-邻近标记技术,从实验原理到实际案例,全面解析这一前沿方法。
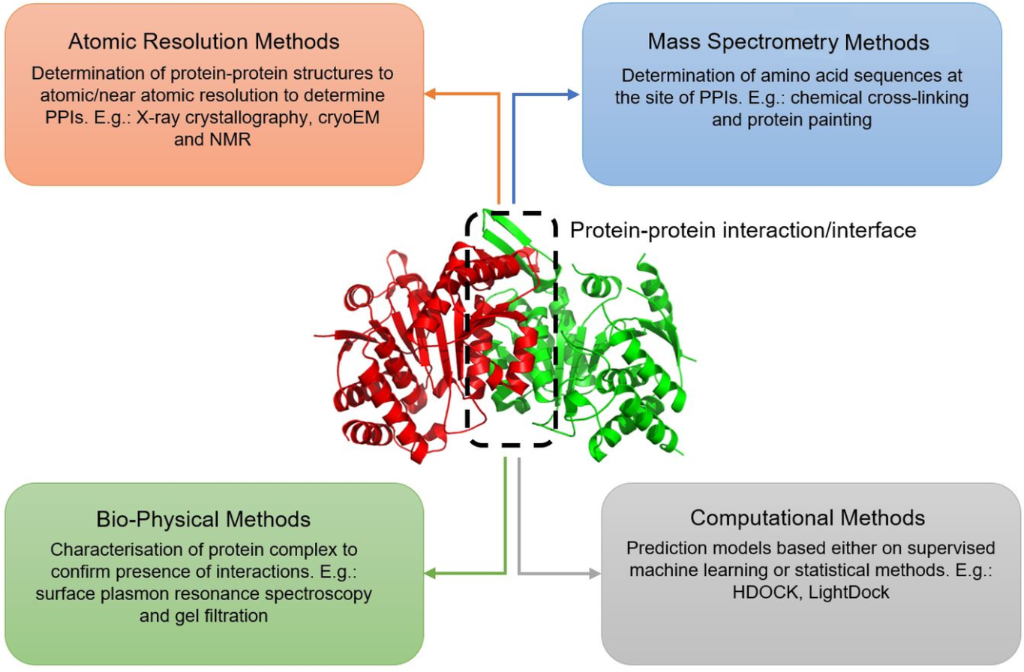
图1研究蛋白互作的相关方法(Seychell et al., 2022)。
PART.1 APEX2邻近标记技术实验原理:
细胞内的"精确制导"系统
APEX2邻近标记技术的核心原理基于工程化的辣根过氧化物酶突变体,通过在活细胞内实现时空精确的蛋白质标记,犹如在细胞内安装了一个"精确制导"系统,能够捕捉蛋白质的瞬时相互作用。
APEX2(增强型辣根过氧化物酶)技术源于APEX,是一种基于工程化辣根过氧化物酶的邻近标记技术。它的发展可追溯到2015年,由Alice Ting实验室在APEX基础上通过定向进化优化而来。APEX2相比前身具有更高的催化活性和稳定性,能够在活细胞中进行实时、高分辨率的蛋白质标记。
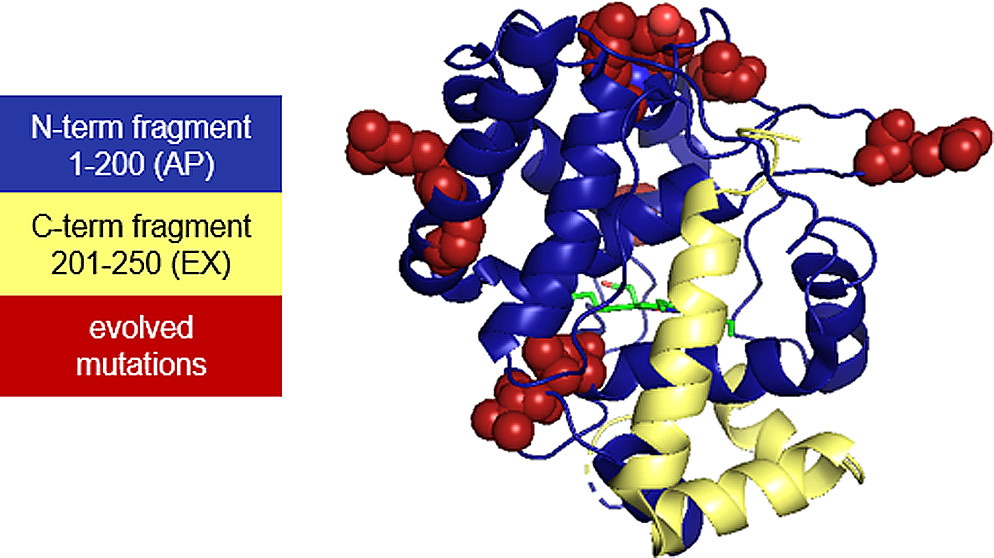
图2 APEX2蛋白的三维结构(Nguyen et al., 2019)。
01 实验基本原理
(1)酶促反应生成活性中间体:当APEX2与底物生物素-苯酚(Biotin-Phenol)和过氧化氢(H₂O₂)共同存在时,APEX2发挥过氧化物酶活性,催化生物素-苯酚生成高反应性的生物素-苯氧自由基(Biotin-Phenoxyl Radical)。这个反应极其迅速,在毫秒级别内完成。
(2)邻近标记过程:生成的生物素-苯氧自由基寿命极短(约1毫秒),扩散距离有限(约20纳米),只能标记APEX2融合蛋白周围纳米范围内的邻近蛋白质。这种空间限制确保了标记的特异性,避免了远距离的非特异性标记。
(3)共价标记与富集鉴定:生物素-苯氧自由基与邻近蛋白质的电子富集氨基酸残基(主要是酪氨酸、色氨酸和半胱氨酸)发生共价结合,实现生物素标记。随后通过链霉亲和素珠进行高效富集,再结合质谱分析鉴定相互作用的蛋白质。
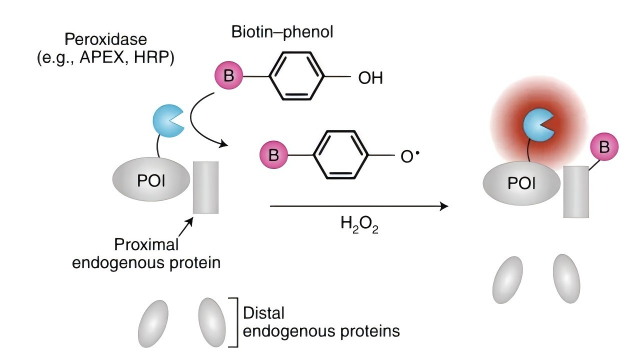
图3 APEX2邻近标记技术原理示意图(Qin et al., 2021)。
02 技术优势
(1)高时空分辨率:整个标记过程可在1分钟内完成,能够捕捉瞬态的蛋白质相互作用;
(2)适用性广:可在活细胞、固定细胞甚至组织样本中应用;
(3)兼容多种细胞区室:通过将APEX2与特定定位序列融合,可靶向不同的细胞器或蛋白质复合物。
PART.2 实验技术流程:
一步步揭开蛋白质互作面纱
APEX2实验流程可分为多个关键步骤,每个步骤都需精细操作以确保结果的可靠性。以下是典型的APEX2实验流程:
(1)载体构建与细胞转染:首先,将APEX2基因与目标蛋白基因融合,构建表达载体。通过质粒转染或病毒转导,将融合基因导入细胞中,确保APEX2定位于目标细胞区室(如线粒体、细胞核等)。优化表达条件至关重要,避免过度表达导致非特异性标记。
(2)生物素-苯酚处理与过氧化氢激活:在细胞培养中加入生物素-苯酚,使其渗透到细胞内。随后,加入低浓度过氧化氢(通常为1 mM)激活APEX2酶反应。反应时间极短(约1分钟),以最小化细胞毒性并确保标记特异性。反应后立即用淬灭缓冲液(如含硫代硫酸钠和抗坏血酸的PBS)终止。
(3)细胞裂解与蛋白质提取:裂解细胞,提取总蛋白质。注意使用温和裂解条件以避免蛋白质降解,并加入蛋白酶抑制剂保护样品完整性。
(4)链霉亲和素富集与洗涤:利用链霉亲和素珠富集生物素化蛋白质。洗涤步骤需严格,使用高盐缓冲液和去垢剂(如SDS)去除非特异性结合,减少背景噪声。
(5)蛋白质消化与质谱分析:将富集的蛋白质进行酶解(如胰蛋白酶消化),然后进行液相色谱-串联质谱(LC-MS/MS)分析。质谱数据通过数据库搜索鉴定蛋白质。
(6)数据分析与验证:使用生物信息学工具分析互作网络,结合GO富集分析验证目标蛋白的功能关联。关键互作蛋白需通过独立实验(如Co-IP)进一步确认。
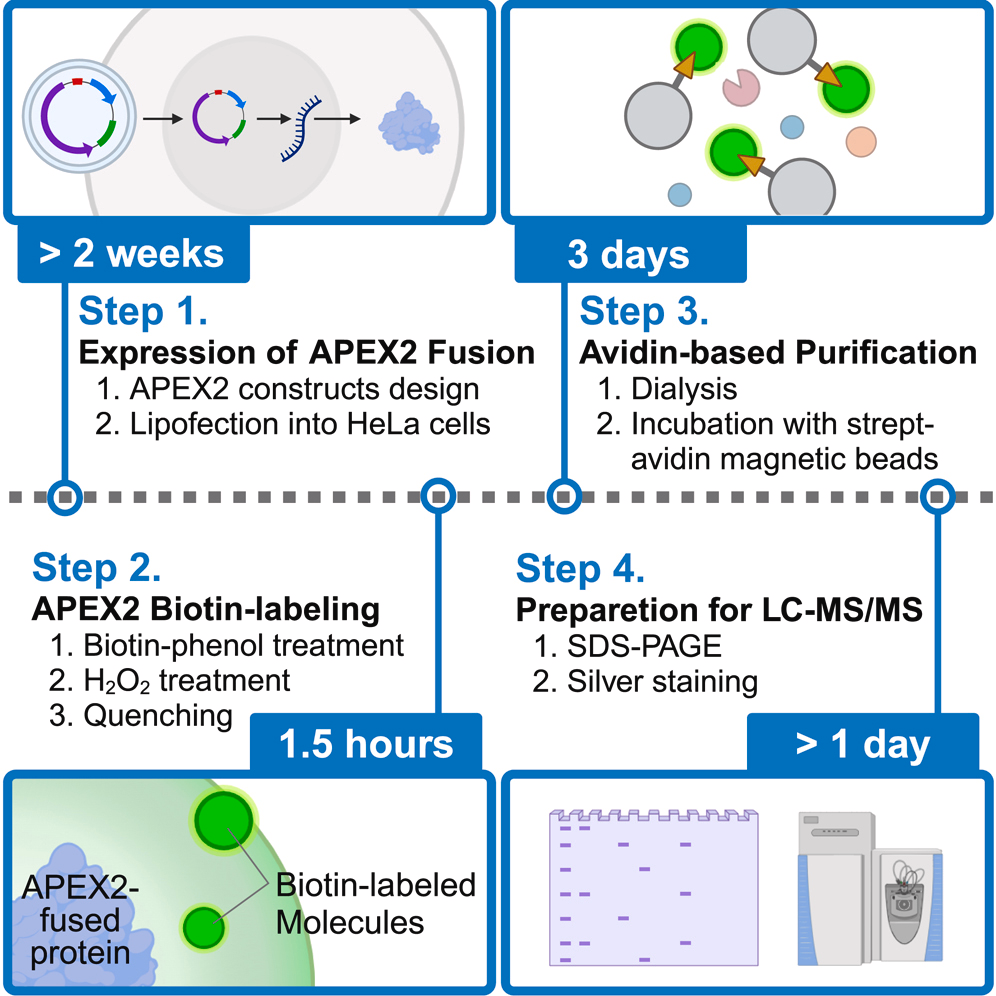
图4 APEX2邻近标记技术的实验流程图(Uozumi et al., 2024)。
PART.3 实验难点与解决方法:
攻克APEX2的挑战
尽管APEX2技术强大,但实验过程中常遇到多个难点,包括标记效率低、背景噪声高和细胞毒性问题。以下是一些常见问题及解决方案:
(1)标记效率低:可能由于APEX2表达不足或反应条件不优。解决方法包括优化转染效率、使用诱导型启动子控制表达,以及调整生物素-苯酚和过氧化氢的浓度与时间。例如,可进行预实验滴定过氧化氢浓度,找到最佳范围(0.5-2 mM)。
(2)高背景噪声:非特异性标记是常见问题,尤其在过表达系统中。可通过设置严格对照(如空载APEX2或突变体)来区分特异性信号。洗涤步骤中,增加高盐洗涤(如1M NaCl)和使用变性缓冲液可有效降低背景。
(3)细胞毒性:过氧化氢可能引起氧化损伤。建议使用短时间反应和低温处理以减少毒性,同时监测细胞存活率。此外,选择适当的细胞类型(如HEK293T或HeLa)可提高耐受性。
PART.4 案例分享:
APEX2-邻近标记的成功应用
为了更直观地理解APEX2技术,我们来看一个实际案例:通过APEX2邻近标记技术揭示了半乳糖凝集素-3在PINK1/Parkin介导的线粒体自噬中的关键作用。
01 研究背景
线粒体自噬是一种选择性清除受损、去极化线粒体的细胞内过程,对于维持细胞稳态至关重要,其功能异常与神经退行性疾病、癌症等多种人类病理状况相关。PINK1/Parkin通路是研究最深入的线粒体自噬途径之一。线粒体受损后,PINK1 kinase稳定并激活,招募E3泛素连接酶Parkin至线粒体,启动清除过程。线粒体外膜破裂(OMM)是该通路中的关键事件,OMM破裂导致线粒体内膜蛋白暴露,从而被自噬机器识别。OMM破裂后发生的分子事件,以及细胞如何"感知"并精确清除这些受损线粒体,尚不完全清楚。
02 实验设计思路
Liu等研究人员将APEX2通过一个柔性连接子融合到PHB2的C末端。PHB2是位于线粒体内膜的蛋白。当加入底物生物素-苯酚和H₂O₂时,激活的APEX2能在其周围约20纳米范围内产生高活性的生物素-苯氧自由基,共价标记邻近的蛋白质。这相当于给PHB2(线粒体内膜)周围的蛋白质环境"拍照"。
用DMSO(对照)或OA(寡霉素/抗霉素A,诱导线粒体自噬)处理表达PHB2-APEX2的HeLa Parkin细胞(Parkin过表达稳转株),然后在特定时间点加入生物素-苯酚和H₂O₂进行短暂标记,以捕获OMM破裂发生时PHB2的邻近蛋白质组。
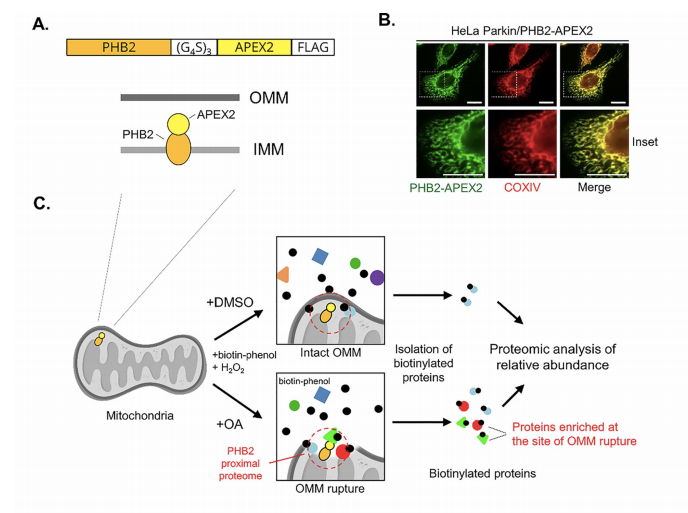
图5 筛选OMM破裂发生时PHB2的邻近蛋白的实验设计思路(Liu et al., 2025)。
03 实验结果
通过免疫荧光染色,显示PHB2-APEX2融合蛋白(绿色)与线粒体标记物COXIV(红色)共定位,证明该融合蛋白成功并正确地定位到了线粒体。链霉亲和素印迹分析显示,只有在表达PHB2-APEX2并加入H₂O₂/生物素-苯酚的细胞中,才出现大量的生物素化蛋白条带。
OA处理后出现了许多独特的条带,表明在线粒体自噬诱导下,PHB2周围的蛋白质环境发生了显著变化。考马斯亮蓝染色显示,从OA处理的细胞中纯化到了更多的生物素化蛋白。
Western Blot进一步验证了纯化组分的特异性:
1)Parkin在基础条件下几乎不存在于PHB2邻近,但在OA处理后显著富集,这与已知的Parkin在线粒体自噬中被招募到线粒体的事实一致。
2)p62/SQSTM1是一个自噬受体,在OA处理后也被富集。
3)Galectin-3作为内膜系统完整性的“哨兵”,在基础条件下检测不到,但在OA处理后显著出现在PHB2的邻近蛋白质组中。
通过精巧的APEX2邻近标记技术,作者成功地"捕捉"到了在线粒体自噬过程中,位于OMM破裂位点、与暴露的IMM蛋白PHB2相互作用的蛋白质网络。从中,他们筛选并验证了Galectin-3作为一个在OMM破裂后特异性被招募到该位点的关键蛋白,为后续深入研究其功能奠定了坚实的基础。
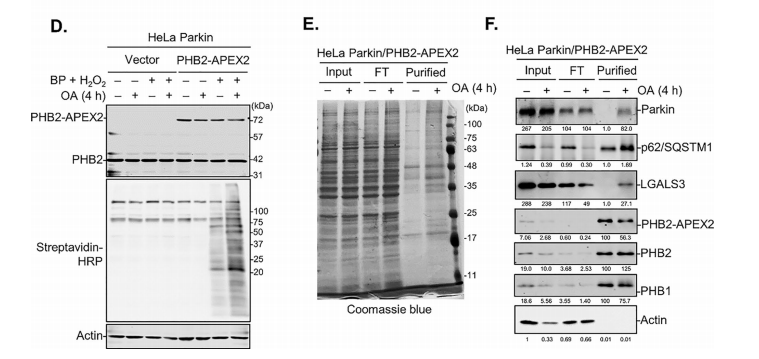
图6 OA处理后,PHB2周围的蛋白质组发生明显变化(Liu et al., 2025)。
参考文献
Liu P H, Lin Y S, Chu W H, et al. Galectin-3 directs mitophagy in response to Parkin-/proteasome-dependent rupture of mitochondrial outer membrane[J]. Biology Direct, 2025, 20(1): 1-21.
Nguyen T M T, Kim J, Doan T T, et al. APEX proximity labeling as a versatile tool for biological research[J]. Biochemistry, 2019, 59(3): 260-269.
Qin W, Cho K F, Cavanagh P E, et al. Deciphering molecular interactions by proximity labeling[J].Nature Methods,2021, 18(2): 133-143.
Seychell B C, Beck T. Molecular basis for protein–protein interactions[J]. Beilstein Journal of Organic Chemistry, 2021, 17(1): 1-10.
Uozumi R, Mori K, Akamine S, et al. Protocols for identifying endogenous interactors of RNA-binding proteins in mammalian cells using the peroxidase APEX2 biotin-labeling method[J]. STAR protocols, 2024, 5(4): 103368.




















 982
982

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








